Databases of biological information
(Minoru Kanehisa)
진행중인 게놈프로젝트에 의해 생산되는 풍부한 생물학 자료들은 생물학자들을 위한 database 도구들을 개발하는데 사용되고 있다. 이 기본적인 생물학 정보는 분자 상호작용부터 생물체간의 상호작용까지 많은 측면에서 풀이될 수 있다.
분자생물학에서 실험적인 기술이 발달하면서 bioinformatics는 생물학의 새로운 분야로 등장하였다. 그 결과 bioinformatics의 주 임무는 자료를 정리하고 관리하며 실험적인 일들에 도움을 주어왔다. 그러나 앞으로의 주요한 임무는 자료들로부터 생물학적 정보를 이끌어내는 일이다. 지금은 대부분 서열과 3-D 구조들이지만 조만간 DNA chip 기술들에 의해 나오는 유전자발현 양상과 같은 다른 형태의 자료들이 이용가능해질 것이다. Bioinformatics에는 대량적인 차원에서 자료해석에 관련된 두가지 측면이 있다: 새로운 algorithm과 소프트웨어들을 개발하고, 자료와 지식을 전산화하는 것이다. 예를 들어 서열들을 사용하여 데이터베이스 검색을 위한 빠르고 정밀한 방법들을 개발하는 것이 중요하긴 하지만 데이터베이스 서열들과 관련된 생물학적 정보의 문헌들이 없이는 결과들의 해석은 불가능할 것이다. 실제로 다른 미확인 서열들과 일치하는 미확인 서열들의 절편은 더많은 완전한 게놈서열들이 데이터베이스에 저장되면서 증가하고 있다. 그러나 자료의 증가량은 반드시 생물학 지식의 증가를 의미하지는 않는다. 생물학적 기능들에 관한 새로운 조작적인 실험들을 고안하는 것은 물론이고 더많은 노력이 생물학 정보들의 데이터베이스를 조직화하는데 기울여져야하는 이유가 바로 여기에 있다.
Sequence-function relationship
잘 갖추어진 문헌, 서열, 3-D 좌표들의 데이터베이스와는 대조적으로 생물학적 기능들의 데이터베이스는 아직 초기 상태에 있다. 생물학 지식을 어떻게 전산화된 데이터베이스들에 나타내고 이용할 수 있는지에 대해서는 논란이 많다. 예를 들어 어떤 데이터베이스는 특정한 분자들이나 기능들에 집중하여 매우 자세한 정보를 제공하는 한편 다른 것들은 다소 상세하지 않은 정보와 함께 생물학의 전반적인 분야를 커버하려고 노력하고 있다. 어떤 데이터베이스들은 사람으로 하여금 읽고 풀이하도록 고안되었고, 어떤 것들은 연역적인 데이터베이스의 형태로 전산화된 추론을 목표로 하고 있다. 어떤 경우에는 생물학적 정보는 다른 데이터베이스들의 전산 분석을 통해 만들어지고; 다른 경우에는 수동으로 문헌으로부터 얻어진다. 다른 시각에서 생물학 정보를 조직화하려고 하는 데이터베이스들을 골라 URL box에 목록을 만들었다.
서열정보와 관련된 기능적인 자료들은 서열 데이터베이스들의 특징을 보여주는 표에 저장되어 있다. 그러나 그 정보는 별로 표준화되어있지 않다. 왜냐하면 주석들이 다른 저자들에 의해 만들어졌기 때문이다. SWISS-PROT는 예외로서 가장 정돈이 잘된 단백질 서열 데이터베이스이다. PROSITE는 SWISS-PROT에서 나온 것으로 특별한 기능들과 관련된 보존된 서열 pattern들 (또는 서열 모티브들)을 포함하고 있다. BLOCKS와 PRINTS들도 모티브 데이터베이스이고, Pfam과 ProDom은 보다 긴 서열들의 pattern들인 단백질 도메인 구조들을 담고 있다. 주요한 차이점들 중 하나는 어떻게 서열정보를 보여주는가 하는 것이다. 즉, text pattern들의 regular expression, multiple alignments, profiles 또는 HMMs. 모티브 도서관들은 단어들의 의미 (subsequences)를 사람이 이해하도록 작성한 외국어 사전과 같다.
Classification of molecules
서열 모티브들에서 나타나는 것처럼 서열들의 국부적인 유사성은 단백질들의 특이적인 기능을 반영한다. 반면 보다 넓은 기능적 위계가 단백질 서열들의 전체적인 유사성과 단백질 입체구조들의 전체적인 유사성에 의해 구축될 수 있다. URL box는 몇가지 대표적인 데이터베이스들을 보여준다: PIR은 단백질 서열들의 초집단 (superfamily) 분류를 포함하고, SCOP와 CATH는 단백질 입체구조들의 위계적 분류 (4단계 수준: classes, folds, superfamilies, and families)이다. 두가지 단백질이 분명한 서열유사성이 없음에도 공통적인 입체구조를 보여줌으로써 동일한 기능적 범주에 속하는 것을 보여주는 일이 자주 있다. 이러한 형태의 정보는 SCOP 같은 곳에서 얻어질 수 있다.
단백질 서열이나 입체구조 전체를 분류하는 일반적인 목적의 데이터베이스와는 대조적으로 특정한 범주의 것에만 특화된 다양한 데이터베이스들이 있다. URL box에서는 이들 중 몇가지만을 보여준다. TRANSFAC은 전사조절인자, GCRDb는 G-protein-coupled receptor들, PKR은 단백질인산화효소에 대한 것이다. 이러한 특화된 데이터베이스들의 대부분은 개인적인 차원에서 운영되기 때문에 질과 양적인 차이가 심하다. Proweb은 현재 이용가능한 특이적인 단백질 site들에 대한 좋은 안내를 제공한다.
Wiring diagrams of molecules
위에 언급된 데이터베이스들은 생명체의 구조물이라고 할 수 있는 개개의 분자들에 관한 생물학적 정보를 담고 있다. 그러나 상호작용하는 분자들의 얽힌 네트워크가 살아있는 세포를 형성한다. 그러한 상호작용들이나 분자들의 배선도 (wiring digram)에 대한 지식이 없이는 세포내 기능들에 대한 이해는 불가능할 것이다.
URL box에 있는 생화학 경로의 데이터베이스들은 특히 대사경로들을 위한 상호작용의 네트워크이다-KEGG와 WIT는 많은 종들의 일반적인 목적의 데이터베이스들이다. EcoCyc는 Escherichia coli에 특이적인 것이고, UM-BBD는 세균에서 이차 생분해에 대한 특수한 데이터베이스들이다. LIGAND는 KEGG와 밀접하게 연결되어 있으며 효소반응과 화학적 성분들에 대한 정보를 담고 있다. 반응이나 상호작용은 배선단위 (a unit of wiring)을 나타내나 효소반응과 같은 것들 이외의 상호작용 데이터베이스들은 아직까지 개발되지 않았다.
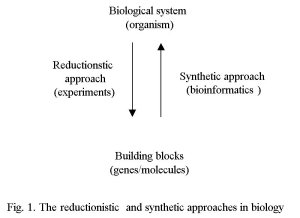
Box 1. 생물체의 재구성 분자생물학에서 궁극적인 환원주의의 궁극적인 형태는 게놈 프로젝트로서 이것은 실험적인 기술로 유전자들의 완전한 세트를 확인하는 것이다 (그림 1 참조). 이것은 bioinformatics에서 새로운 도전을 부여한다. 완전한 게놈서열이 주어진다면 생물체의 기능적인 체계를 재구성하는 것이 가능한가? 단지 게놈서열만이 주어진다면 그 해답은 'no'이다. 그러나 만약 단백질 상호작용들 (yeast two-hybrid system에 의해 확인되는)과 유전자 상호작용들 (유전자 붕괴나 과발현 후에 발현양상에 의해 확인되는)에 대한 체계적인 실험자료가 추가된다면 그 전망은 밝다. 따라서 우리는 서열정보를 가진 완전한 유전자들, 불완전한 단백질과 유전자상호작용, 그리고 KEGG 같은 데이터베이스에 저장되어 있는 분자배선도들의 불완전한 참고지식이 주어져 있다. 재구성을 위한 일반적인 규약은 우선 참고물들의 배선도를 만들고 부분적인 subsystem들을 구축하고, 그리고 빈자리를 메꾸어 system의 전체적인 그림을 얻는 것이다. 성공은 유전학, 생화학, 분자와 세포생물학에서의 관련된 실험들의 질과 양뿐만이 아니라 이 실험들을 포함하는 참고 데이터베이스의 질과 완성도에 달려있다. Bioinformatics는 참고 데이터베이스를 개발하고, 분자적 배선을 예측하기 위해 체계적인 실험을 고안하고 자료를 처리하는데 있어 중요한 역할을 할 것이다. |
경로에 관한 데이터베이스들은 게놈 프로젝트에서 나오는 자료들을 체계적으로 처리하기 위하여 점차적으로 중요성이 증가하고 있다. 예를 들어 KEGG에서는 기능적 단위체들이 정확히 형성되는지 점검하기 위해 유전자들의 일람표가 서열유사성에 의해 관련된 경로들에 표시될 수 있다. 많은 생물체들의 전체 게놈분석의 결과로 서열 데이터베이스들이 빠르게 팽창하고 있는 것과 마찬가지로 경로 데이터베이스들도 기능게놈학 (functional genomics)에서의 체계적인 실험들을 따라 팽창하도록 되어있다 (Box 1 참조).
|
Comparison and classification of organisms
완전한 게놈서열들의 이용은 다른 종으로부터의 전체 유전자들의 비교분석을 가능하게 해주고 있다. 이러한 분석들의 결과들은 orthologous gene들의 묶음으로 표시되어 KEGG의 ortholog 그룹에는 물론이고 COG와 MBGD와 같은 데이터베이스에 저장된다. 이러한 데이터베이스들에 있는 자료들은 유전자들의 multiple alignment를 반영하기 때문에 이들에 대한 유사성 검색은 일차적인 서열자료들보다 훨씬 믿을 만 하다. 그러나 이런 방식으로 확인될 수 있는 유전자 집단들은 현재로서는 그리 크지 않다.
형태적, 분자적 증거에 근거하여 생물체를 분류하는 것은 어렵고 논쟁의 여지가 있다. URL box는 그러한 정보를 담고있는 두 개의 site를 보여준다 - NCBI Taxonomy 데이터베이스는 GenBank 데이터베이스에 적용된 생물체들의 분류를 담고있으며, Tree of Life는 계통수적 상관관계와 생물체들의 특징들을 총괄하여 놓았다. Comparative genomics에 의해 수집되는 정보가 팽창하는 것을 볼 때 생물체 분류에 관한 데이터베이스들도 상당한 혜택을 보게될 것이다.
Future directions
Bioinformatics는 생의학 분야의 기초연구만이 아니라 응용연구에서도 필수적인 도구가 되었다. URL box에는 몇군데 의학 데이터베이스들을 추려놓았다. OMIM은 인체 유전자들과 유전질환들의 목록을 제공한다; Merck Manual은 임상정보에 대한 자료를 제공한다. 일반적으로 의학 데이터베이스들은 사람이 검색하여 읽을 수 있는 교과서와 같아서 현존하는 분자생물학 데이터베이스들의 Web과 연계가 부족하다. 그렇지만 더많은 질환유전자들이 밝혀지고 oligonucleotide chip들이 그들의 돌연변이 유형을 축적하게 되면 상황이 변할 것이다.
Bioinformatics는 대량의 서열자료생산 시대의 생물과학과 컴퓨터과학 간의 결혼에서 탄생하였다. 이 결혼은 성공적이었지만 게놈학에서의 진보는 상호작용과 돌연변이들에 대한 새로운 형태의 자료들을 생산하기 시작할 것이다. 동시에 단지 문헌이나 장인의 머리 속에 있는 분자적 상호작용에 대한 많은 양의 지식이 있다. 따라서 생물학적 정보의 데이터베이스들의 발달에 많은 진전이 기대된다.
URLs |
|||||
Sequence annotation |
SWISS-PROT |
http://expasy.hcuge.ch/sprot/ |
Biochemical pathways |
KEGG |
http://www.genome.ad.jp/kegg/ |
Sequence motifs |
PROSITE |
http://expasy.hcuge.ch/sprot/prosite.html |
|
LIGAND |
http://www/genome.ad.jp/dbget/ligand.html |
|
Blocks |
http://www/blocks.fhcrc.org/ |
|
WIT |
http://www.cme.msu.edu/WIT |
|
PRINTS |
http://www.biochem.ucl.ac.uk/bsm/dbbrowser/PRINTS/ |
|
EcoCyc |
http://ecocyc.PangeaSystems.com/ecocyc/ |
|
Pfam |
http://www.sanger.ac.uk/Pfam http://pfam.wustl.edu/ |
|
UM-BBD |
http://www.labmed.umn.edu/umbbd/ |
|
ProDom |
http://protein.toulouse.inra.fr/prodom.html |
Orthologous genes |
COG |
http://www.ncbi.nlm.nih.gov/COG/ |
Sequence classification |
PIR Superfamily |
http://www-nbrf.georgetown.edu/pir/ |
|
MBGD |
http://mbgd.genome.ad.jp/ |
3-D fold classification |
SCOP |
http://scop.mrc-lmb.cam.ac.uk/scop |
Organism classifications |
NCBI Taxonomy |
http://www.ncbi.nlm.nih.gov/Taxonomy/ |
|
CATH |
http://www.biochem.ucl.ac.uk/bsm/cath |
|
Tree of Life |
http://phylogeny.arizona.edu/tree/phylogeny.html |
Specific molecules |
TRANSFAC |
http://transfac.gbf-braunschweig.de/TRANSFAC/ |
Medical resources |
OMIM |
http://www.ncbi.nlm.nih.gov/Omim/ |
|
GCRDb |
http://www.gcrdb.uthscsa.edu/ |
|
HGMD |
http://www.uwcm.ac.uk/uwcm/mg/hgmd0.html |
|
PKR |
http://www.sdsc.edu/kinases/ |
|
Merck Manual |
http://www.merck.com/pubs/mmanual/html/sectoc.htm |
|
Proweb |
http://www.proweb.org/ |
|
|
|